Los datos
En este tutorial usaré los conjuntos de datos del Rice SNP-Seek Database, el cual contiene información sobre genotipos de SNP y fenotipos de distintas variedades de arroz (Oryza sativa L.).
Descripción de los datos
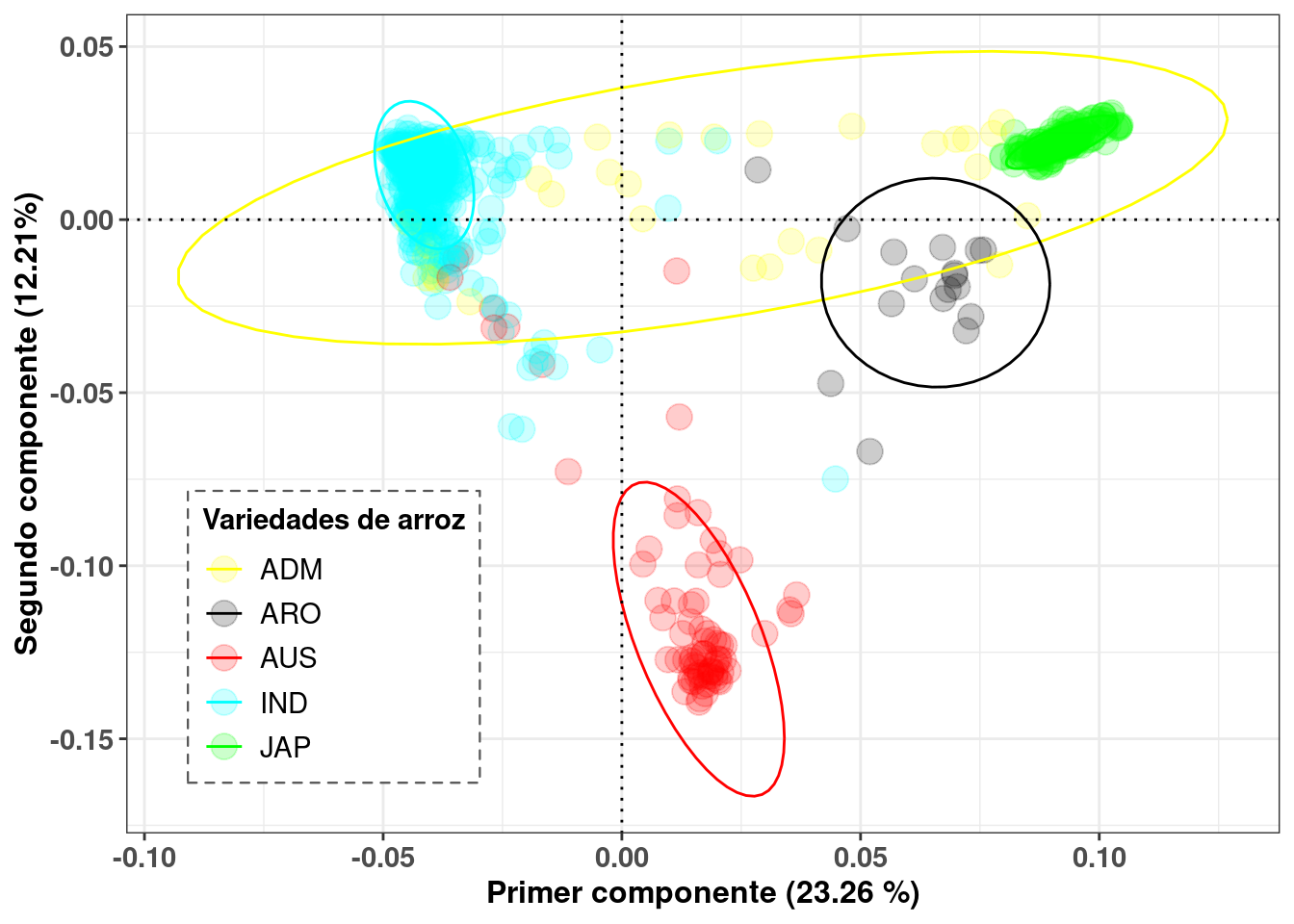
Al realizar un análisis de componentes principales sobre los datos de genotipo, se observaron diferentes grupos varietales de arroz (Figure 1). La variedad indica fue la variedad seleccionada para llevar a cabo el estudio ya que fue el grupo varietal con mayor número de individuos genotipados (451 individuos de un total de 738).
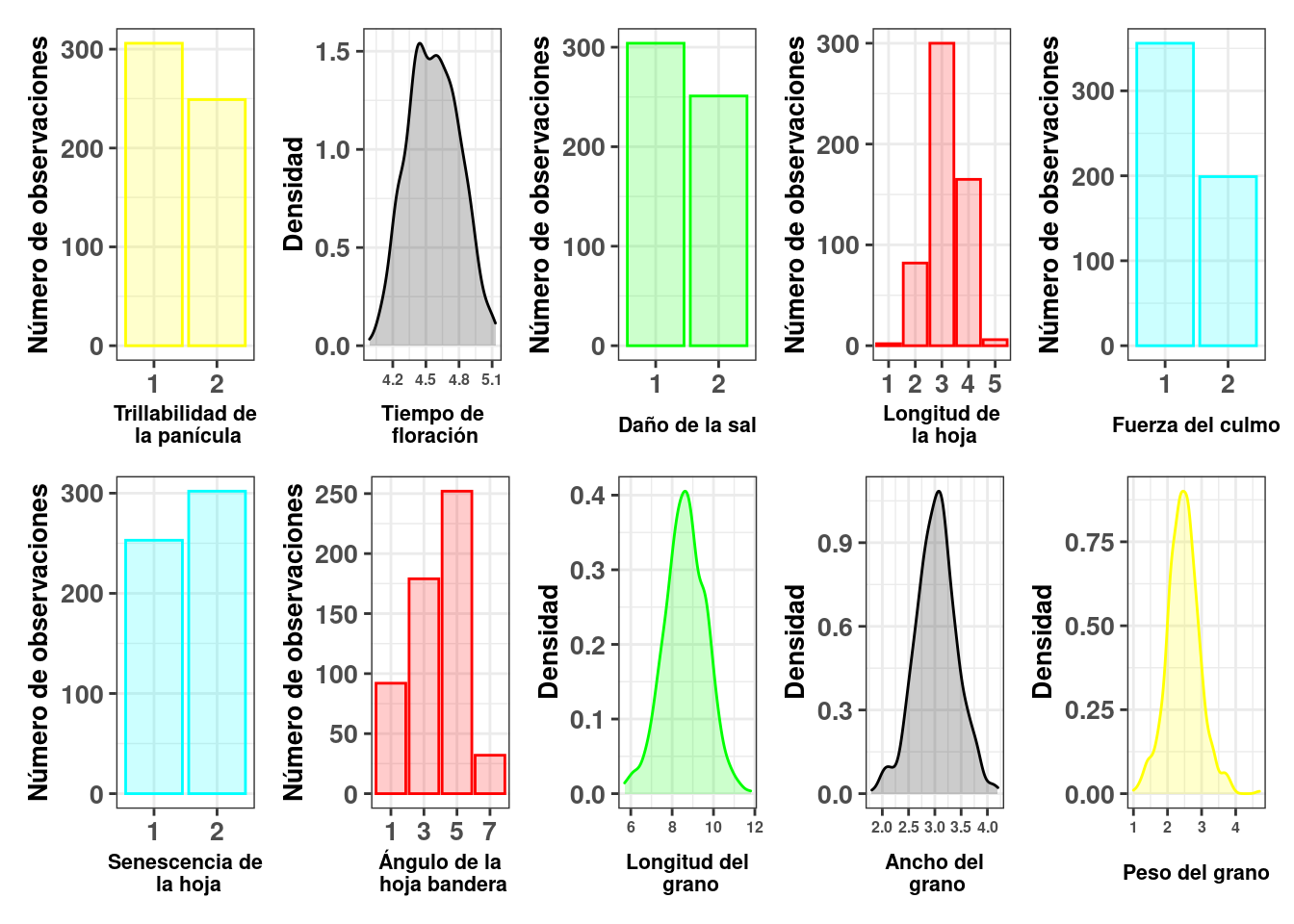
De entre los datos de fenotipos disponibles (Figure 2), se eligió el carácter tiempo de floración, ya que se observó que en este carácter la predicción genómica podría funcionar mejor. Estos datos fenotípicos se centraron (restando la media general) y estandarizaron (dividiendo por la desviación estándar).
Control de calidad
Los datos fueron sometidos previamente a procedimientos de control de calidad, eliminando loci con una frecuencia del alelo menor inferior a 0.05 y con una tasa de ausencia mayor a 0.01, todo esto usando Plink (Purcell et al. 2007).
Luego de dicho control de calidad, el conjunto de datos resultante consistió de 100.231 SNP. A continuación podrá descargar los datos en formato variant call luego del control de calidad.
Antes de realizar cualquier tipo de análisis, se debe verificar la calidad de los datos de genotipo. A esto se le conoce como control de calidad. Uno de los procedimientos de control de calidad que se llevó a cabo fue eliminar SNP de acuerdo a su tasa de ausencia, esto es, SNP que no están bien genotipados en toda la población. El otro procedimiento de control de calidad que se llevó a cabo fue la llamada frecuencia alélica menor, que es la proporción de ocurrencia del alelo que se presenta con menor frecuencia. Un umbral típico para ambos tipos de control de calidad oscilan entre 1 y 5%.